Un equipo del CSIC analiza por primera vez a gran escala cómo se adapta la bacteria del estafilococo a los seres humanos
El Instituto de Biomedicina de Valencia (CSIC) colidera un estudio que describe la adaptación genética de la bacteria Staphylococcus aureus durante su colonización en personas portadoras
El Instituto de Biomedicina de Valencia (CSIC) colidera un estudio que describe la adaptación genética de la bacteria Staphylococcus aureus durante su colonización en personas portadoras
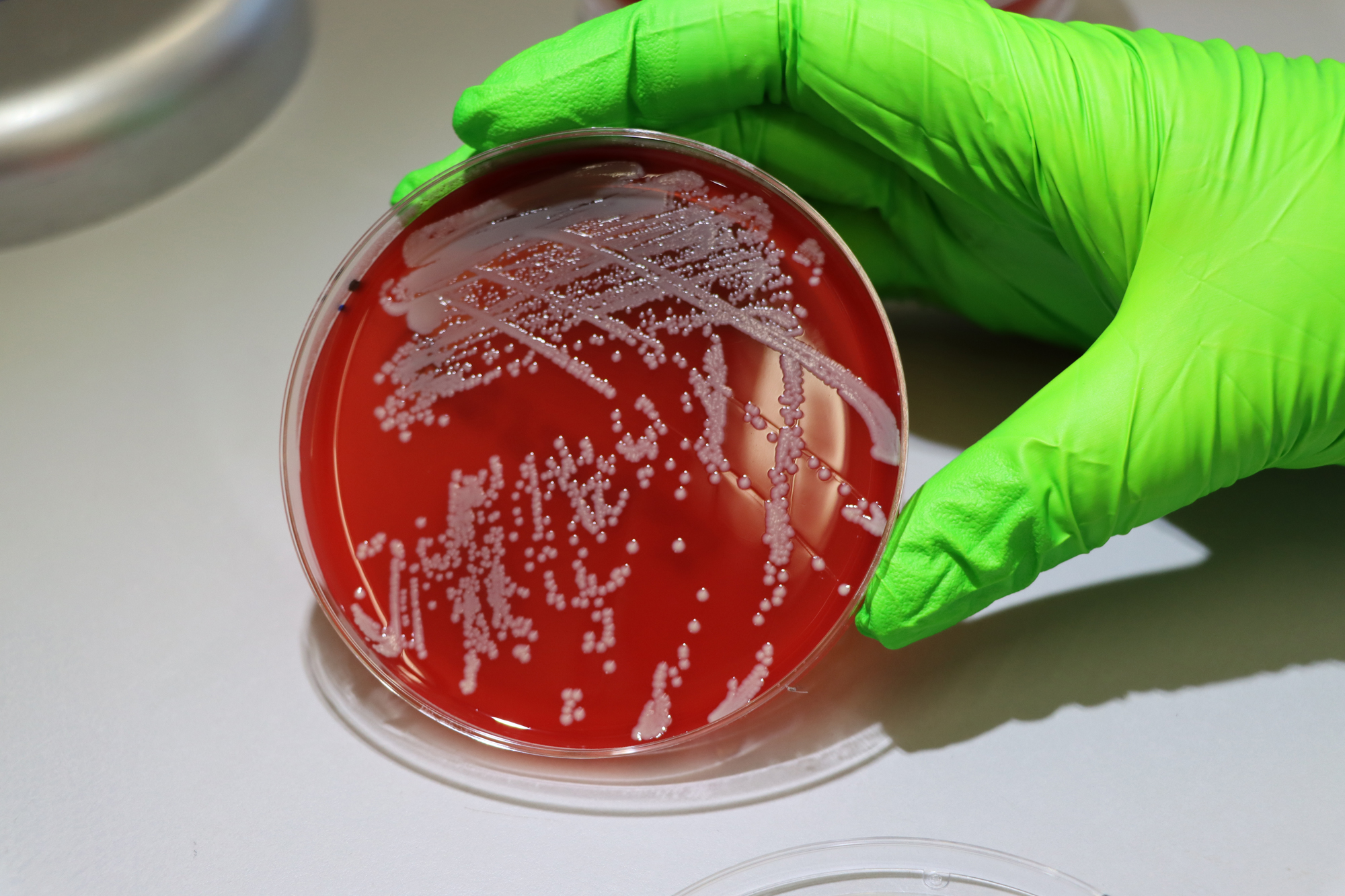
Un equipo internacional coliderado desde el Instituto de Biomedicina de Valencia (IBV), del Consejo Superior de Investigaciones Científicas (CSIC), publica hoy en Nature Communications el estudio más detallado hasta la fecha sobre los mecanismos por los que el tipo más común de bacteria del estafilococo, Staphylococcus aureus, se adapta a vivir en el cuerpo humano. Presente en el 30% de la población, principalmente en la microbiota de la piel y el intestino, esta bacteria es inofensiva para la mayoría de las personas, aunque en determinadas circunstancias puede causar infecciones graves. Este estudio, realizado por primera vez mediante análisis genéticos de la bacteria a partir de muestras de personas portadoras, podría ayudar a mejorar la prevención, el diagnóstico y los tratamientos de las infecciones causadas por estos microorganismos.
El estafilococo forma parte de la microbiota humana, el conjunto de bacterias que habita en la piel, el intestino, la boca y el tracto respiratorio superior, donde se encuentra en alrededor del 30% de la población, habitualmente en la piel y la nariz. La bacteria más común de esta familia, Staphylococcus aureus (cuyo nombre hace referencia al aspecto que tiene al microscopio, como un “racimo de uvas doradas”), es inofensiva para la mayoría de las personas, aunque en determinadas circunstancias puede causar desde infecciones cutáneas, las más comunes, hasta infecciones invasivas del torrente sanguíneo más raras (sepsis). Las infecciones son más graves en personas con otras afecciones de salud.
“En este estudio, hemos analizado los genomas de más de 7.000 muestras de Staphylococcus aureus obtenidas de más de 1.500 portadores humanos para identificar cambios genéticos que se originaron en la bacteria en su huésped y entorno natural. El uso de un análisis computacional nos permitió identificar los cambios genéticos en esta bacteria que probablemente contribuyen a una mejor supervivencia y permanencia durante la colonización humana”, explica Francesc Coll, científico titular del CSIC en el IBV y autor principal del trabajo que publica hoy Nature Communications.
Los autores, entre los que hay científicos de las universidades de Cambridge, Birmingham y Bristol (Reino Unido), y del Trinity College y la Universidad de Cork (Irlanda), adoptaron un enfoque experimental novedoso: en lugar de observar la adaptación bacteriana en el laboratorio, analizaron los genomas de S. aureus de portadores humanos para identificar cambios genéticos recurrentes.
“Nuestro estudio ha permitido estudiar por primera vez a gran escala la adaptación genética de S. aureus durante su colonización en personas portadoras. Los estudios previos se centraron en investigar su adaptación en condiciones de cultivo de laboratorio, o en casos de infección en humanos”, revela el investigador.
Metabolismo del nitrógeno y resistencia a antibióticos
Aunque interpretar las condiciones exactas a las que se adaptaron las bacterias no siempre es sencillo, este método proporcionó una forma indirecta de descifrar cómo las bacterias sobreviven y se adaptan en su huésped y en su entorno natural. Los autores identificaron por primera vez cambios en genes asociados con el metabolismo del nitrógeno, “lo que sugiere que se trata de un proceso metabólico clave necesario para la colonización de humanos por S. aureus”, asegura Coll. El estudio también identifica mutaciones en genes que podrían influir en la forma en que la bacteria interactúa con las células humanas y el sistema inmunológico.
Así, el trabajo mostró que, en algunos casos, S. aureus desactiva el sistema de regulación que controla los factores y las toxinas que contribuyen a la virulencia durante las infecciones. “Esto podría representar una estrategia de evasión del sistema inmunológico, o una forma para que algunas células de S. aureus se beneficien de los factores de virulencia secretados por otras células sin tener que producirlos ellas mismas, lo que llamamos células ‘tramposas’”, argumenta el investigador del CSIC.
Además, el estudio comprobó que S. aureus adquiere mutaciones de resistencia a antibióticos como el ácido fusídico, la mupirocina y la trimetoprima, demostrando que estas mutaciones efectivamente confieren resistencia a estos antibióticos en el laboratorio. La Organización Mundial de la Salud (OMS) considera las bacterias resistentes a antibióticos como uno de los problemas más graves a los que se enfrenta la humanidad en el futuro próximo, e incluye a Staphylococcus aureus resistente a la meticilina en su lista de patógenos bacterianos prioritarios de 2024.
Mejorar la prevención y tratamiento de las infecciones
“En conjunto, este estudio revela procesos biológicos clave que S. aureus emplea para sobrevivir y persistir como bacteria comensal en humanos”, resume Francesc Coll. Así, el estudio de la evolución y adaptación genética de las bacterias en su entorno natural, ya sea durante la colonización asintomática de sus portadores o en el establecimiento y transcurso de infecciones, puede ayudar a mejorar la prevención, el diagnóstico y los tratamientos de las infecciones, aseguran los autores.
“Comprender cómo responden las bacterias a los tratamientos con antibióticos ha permitido identificar los cambios genéticos que les permiten sobrevivir al ataque de los antibióticos”, recuerda Coll. Estas mutaciones pueden utilizarse como marcadores diagnósticos, así como para diseñar nuevas estrategias terapéuticas y un uso más racional y eficaz de los antibióticos. Estudios de la adaptación bacteriana como este también podrían revelar mecanismos de evasión inmunológica, cómo las bacterias se adaptan para eludir el reconocimiento y ataque del sistema inmune. “Esto podría ayudar a identificar nuevos antígenos, componentes de la bacteria que el sistema inmunitario reconoce como extrañas o peligrosas, y diseñar nuevas vacunas”, puntualiza.
CSIC Comunitat Valenciana
Coll F, Blane B, Bellis K, et al. The mutational landscape of Staphylococcus aureus during colonisation. Nature Communications. 2024. DOI:10.1101/2023.12.08.570284v1
Noticias relacionadas
Un estudio internacional publicado en la …
Un estudio del Instituto de Gestión de la Innovación y del Conocimiento (…
Las comunidades bacterianas naturales son esenciales para la vida en la…