- Estado de desarrollo
-
Validación preliminar con muestras clínicas
- Propiedad industrial
-
PCT solicitada
- Colaboración Propuesta
-
Licencia y/o codesarrollo
- Solicitud de información
-
Xavier GregoriVicepresidencia de Innovación y Transferenciaxavier.gregori@csic.escomercializacion@csic.es
- Referencia
-
CSIC/XA/005
Información adicional
#Salud
#Diagnóstico
#Biomarcador
#Enfermedades infecciosas
Método mejorado para la detección de patógenos respiratorios virales de RNA
Método de amplificación de señal para la detección de virus respiratorios de RNA en muestras complejas. La combinación de la tecnología de balizas moleculares y la digestión enzimática ha demostrado ser una alternativa altamente específica, rentable y rápida para detectar patógenos víricos.
- Necesidad del Mercado
-
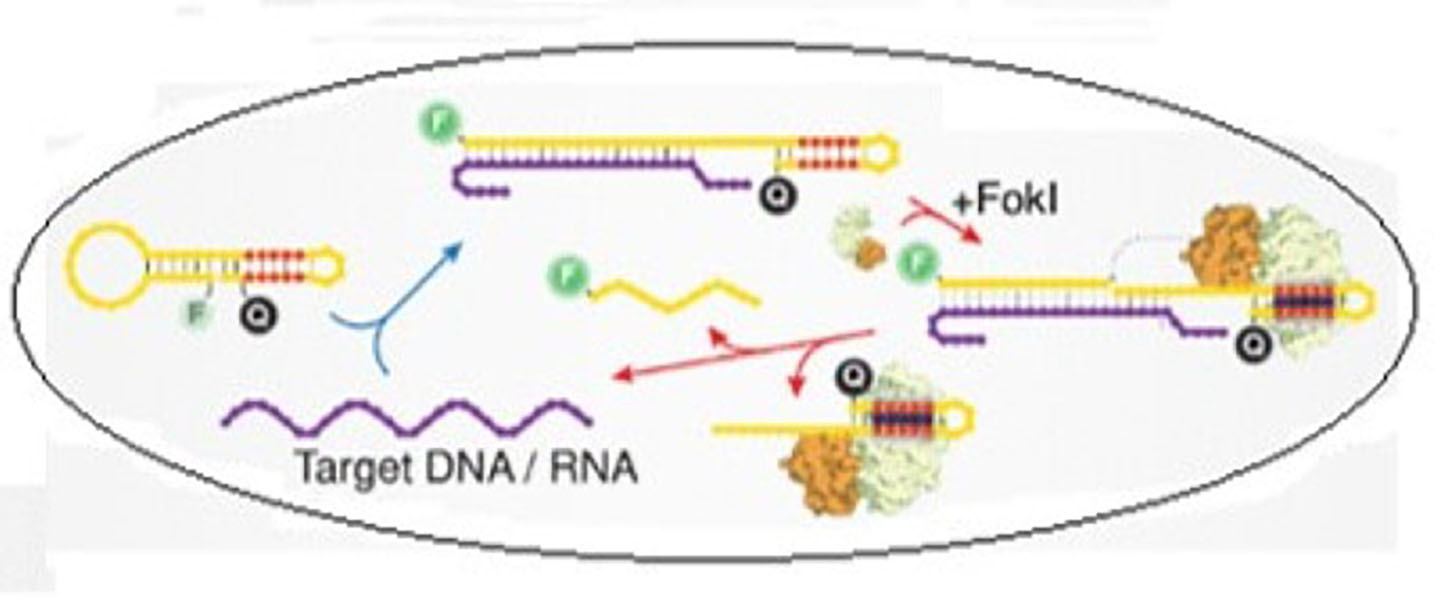
El diseño de nuevas tecnologías de detección directa de RNA, basadas en el aumento de señal que eviten los inconvenientes derivados de los procedimientos de amplificación de ácidos nucleicos al analizar muestras complejas, sigue siendo un reto. Esto es especialmente relevante para la identificación diferencial de virus respiratorios. En este sentido, el uso de la enzima de restricción Fokl, que corta híbridos DNA:RNA, combinado con el uso de balizas moleculares específicas (sondas marcadas con fluorescencia) parece ser una alternativa adecuada.
- Solución propuesta
-
Se ha desarrollado un método en el que, tras la unión del material diana a una baliza molecular específicamente diseñada, la digestión con Fokl libera un grupo fluoróforo, amplificando exponencialmente la señal debido a la recirculación de la secuencia diana. Esta estrategia reduce considerablemente los límites de detección de los RNA diana, con una alta especificidad independientemente de la complejidad de las muestras. Además, presenta capacidades multiplex, ya que se pueden detectar simultáneamente diferentes secuencias virales en la misma muestra. Entre los patógenos virales respiratorios que se han analizado se incluyen Betacoronavirus, SARS-CoV-2, Parainfluenza y Pneumovirus.
- Ventajas competitivas
-
- El límite de detección se ha reducido más de 3 veces en muestras de RNA en comparación con los ensayos de hibridación convencionales.
- Proporciona una alta especificidad y capacidades multiplex, siendo capaz de discriminar, en un único ensayo combinado, un conjunto de patógenos virales respiratorios de interés.
- Mejora el límite de detección de moléculas diana entre 2 y 4 órdenes de magnitud cuando se combina con amplificación isotérmica.