- Tipo de expresión:
- Doctorado: Propuesta de dirección de tesis doctoral/temática para solicitar ayuda predoctoral ("Hosting Offer o EoI")
- Ámbito:
- Química, Biología Estructural, Interacciones Biomoleculares
- Área:
- Materia
- Modalidad:
- Ayudas para contratos predoctorales para la formación de doctores (antiguas FPI)
- Referencia:
- 2023
- Centro o Instituto:
- INSTITUTO DE INVESTIGACIONES QUIMICAS
- Investigador:
- JESUS ANGULO ALVAREZ
- Palabras clave:
-
- RMN, STD NMR, Reconocimiento Molecular, Interacción Ligando-Proteína, Carbohidratos, Estructura, Dinámicas Moleculares, Modelado Molecular, 19F NMR
- Documentos anexos:
- 606639.pdf
- 606640.pdf
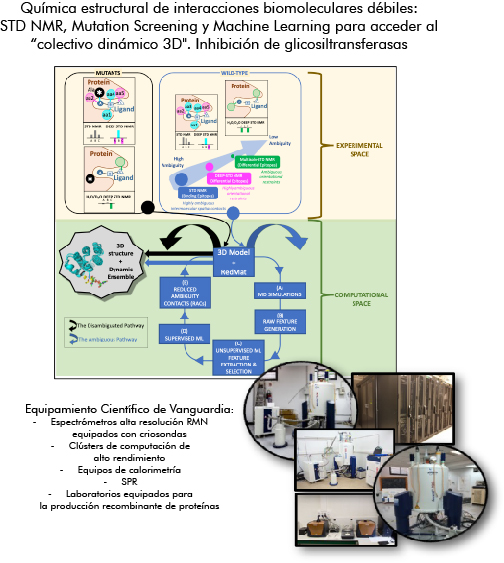
PRE2023 - "Quimica estructural de interacciones biomoleculares débiles: STD NMR, Mutation Screening y Machine Learning para acceder al colectivo dinámico 3D". Inhibición de glicosiltransferasas" (PID2022-142879NB-I00)
El estudio de complejos proteína-ligando débiles desafía las técnicas biofísicas actuales como la cristalografía de rayos X o la espectroscopía RMN de proteínas, por la baja afinidad de interacción. Sin embargo, los complejos biomoleculares débiles son esenciales para comprender la dinámica y la flexibilidad de respuestas celulares. En el descubrimiento de fármacos, el cribado inicial suele identificar ligandos débiles, por lo que determinar sus estructuras 3D puede resultar difícil, lo que obstaculiza el desarrollo de fármacos más eficaces. Se desarrollarán aquí nuevas formas de determinar estructuras 3D precisas de complejos débiles, mediante RMN sin necesidad de observar la proteína. La metodología propuesta se basa en STD NMR. El grupo de investigación cuenta con un amplio historial de aplicación de STD NMR a sistemas biomoleculares relevantes y ha sido pionero en el desarrollo de enfoques novedosos para estudiar este tipo de sistemas (DEEP-STD NMR, multisolvent-STD NMR, IL-STD NMR). La solución innovadora utilizará mutaciones en aminoácidos del sitio de unión de la proteína para generar nuevas restricciones experimentales denominadas Perturbaciones de Epítopos por Mutaciones (EPMs), así como Machine Learning para generar restricciones teóricas en combinación con epítopos experimentales de los ligandos que denominamos Contactos de Ambigüedad Reducida (RACs). La implementación de estas restricciones en cálculos MD permitirá definir de forma precisa la estructura 3D.
Información adicional
Contactar con la unidad